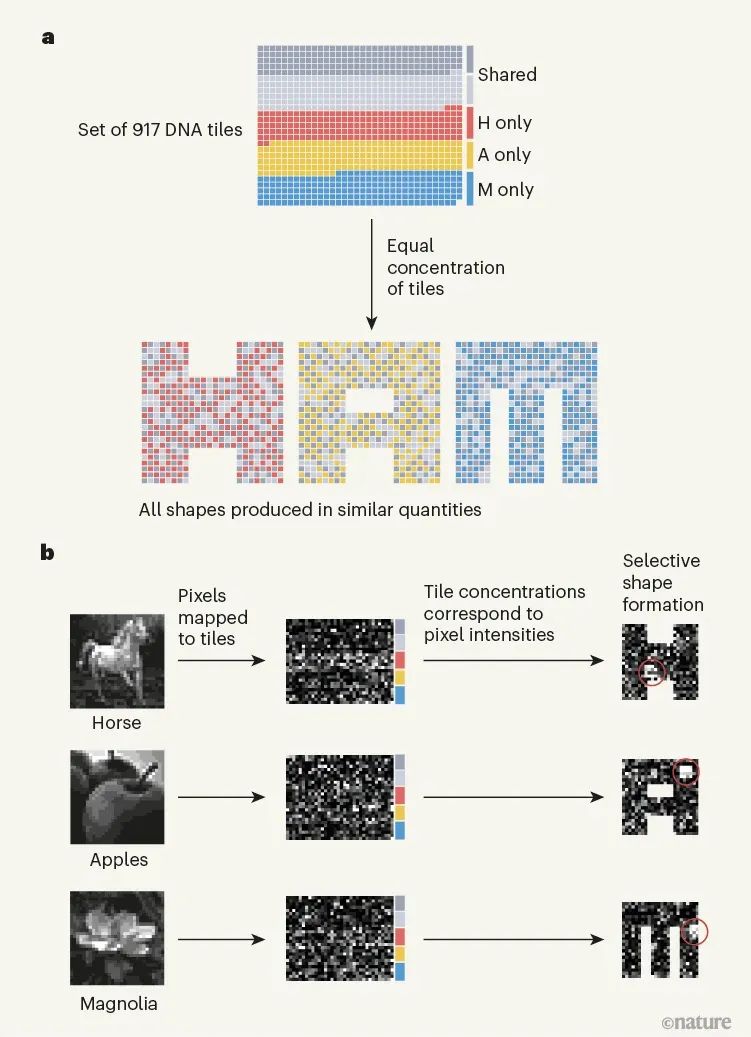
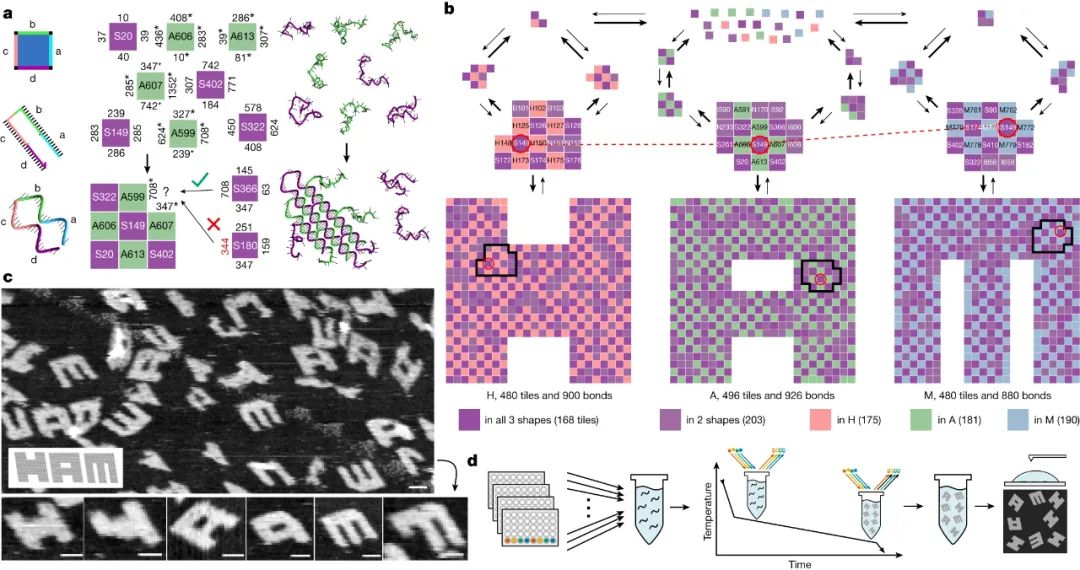
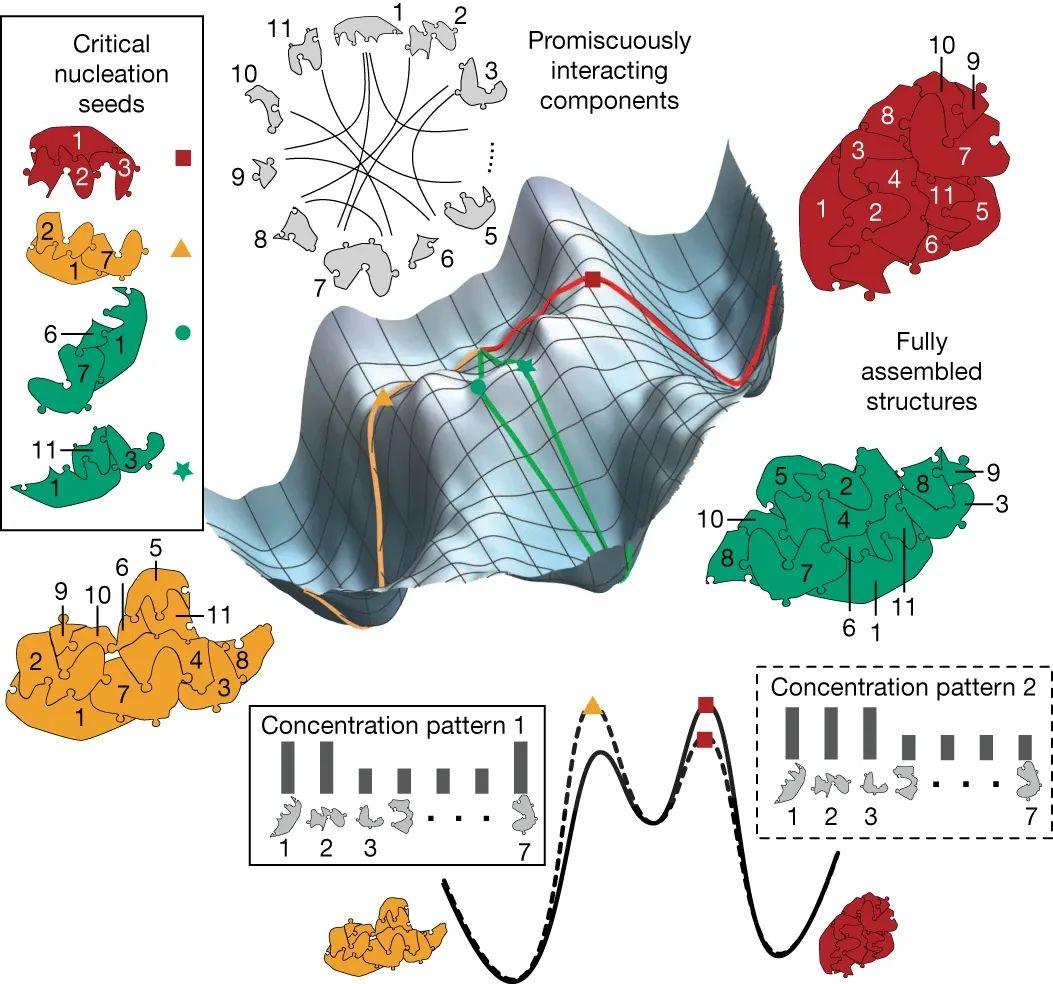
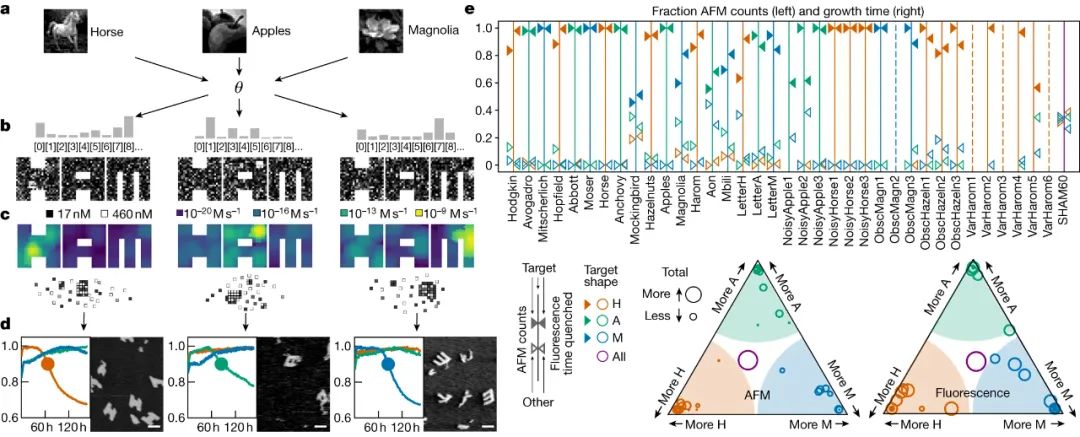
生物系統(tǒng)是如何處理信息的?這樣的計算發(fā)生在多個尺度上,涉及分子、細胞、組織、器官等。例如,計算電路嵌入分子通路中,由大腦的神經(jīng)元形成,免疫系統(tǒng)通過處理大量分子輸入來計算免疫反應(yīng)。了解這種信息處理可以解鎖治療疾病的策略,并確定生物學(xué)的基本工程原理。在人類工程系統(tǒng)中,計算通常在專用的處理單元中進行。相比之下,蛋白質(zhì)合成、分子自組裝和細胞導(dǎo)航等生物過程中涉及的計算往往直接嵌入生物物理過程。理解這種生物物理嵌入的計算仍然是一個關(guān)鍵的挑戰(zhàn)。鑒于此,芝加哥大學(xué)Arvind Murugan、Jackson O’Brien、加州理工學(xué)院Erik Winfree、Constantine Glen Evans等人設(shè)計了一種自組裝DNA系統(tǒng)來進行復(fù)雜的模式識別。作者的工作建立在DNA納米技術(shù)領(lǐng)域的基礎(chǔ)上,該技術(shù)使用DNA構(gòu)建復(fù)雜的納米結(jié)構(gòu),用于廣泛的應(yīng)用。它利用了這樣一個事實,即單鏈DNA可以通過與具有互補序列的DNA分子結(jié)合而在溶液中自組裝。一種策略是以正方形tile的形式從DNA構(gòu)建塊組裝結(jié)構(gòu),正方形tile的設(shè)計使具有互補序列的tile邊緣相互結(jié)合。這種自組裝tile系統(tǒng)已被設(shè)計用于實現(xiàn)廣泛的數(shù)字算法,但尚未利用這些系統(tǒng)探索其他復(fù)雜的計算,如模式識別。為了實現(xiàn)復(fù)雜的模式識別計算,研究人員設(shè)計了一組方形DNA tile,將其自組裝成三種不同的形狀:字母H、A和M。其中一些tile僅用于組裝其中一種形狀;其他的則有兩三種形狀。tile組的設(shè)計使得當(dāng)不同的tile以相等的濃度存在于溶液中時,三種形狀以相似的量形成。相比之下,每種tile的不同濃度導(dǎo)致了一種主要形狀的形成,這取決于濃度。全tile系統(tǒng)是通過計算設(shè)計的,這個過程從三個形狀中的每一個都開始,這些形狀完全由該形狀特有的tile組成,每個tile都有一組不同的邊。然后,合并算法逐漸識別出可以用于多個形狀的tile。該算法的每一步都從兩個不同的形狀中隨機挑選兩個tile,并試圖找到一個可以同時替換它們的tile,從而相應(yīng)地改變相鄰的邊緣。只有當(dāng)生成的系統(tǒng)滿足某些條件時,才能接受更換,以防止系統(tǒng)“卡”在不正確的程序集中。這些條件假設(shè),綁定在單個邊緣上的tile很容易從自組裝形狀脫落,而綁定在多個邊緣上的tile是穩(wěn)定的。當(dāng)滿足條件時,不正確的tile只能在單個邊緣上綁定到系統(tǒng);如果一個不正確的tile確實綁定了,那么隨后的不正確tile也只能綁定在一條邊上。經(jīng)過算法的多次迭代后,獲得了一個優(yōu)化的系統(tǒng),由 917 個tile組成,其中 371 個tile用于多個形狀,具有 698 個不同的邊緣。圖|917種分子的多種混合物,可以從一組分子中組裝成三種不同的結(jié)構(gòu)下一個挑戰(zhàn)是識別僅位于三個組裝形狀之一中的tile組,以形成該形狀的核心。當(dāng)其中一個核心在溶液中形成時,它被稱為成核種子,并且其余形狀的組裝會以犧牲其他兩個形狀為代價而加速——這是“贏者通吃”的結(jié)果。這意味著可以通過選擇特定的tile濃度組合(稱為濃度模式)來控制從全套tile中自組裝的形狀,該組合可以促進該形狀的成核種子的形成。研究人員使用一種算法來估計不同濃度模式形狀的自組裝率,從而識別出 37 個成核種子。然后使用 150 小時的退火工藝對相應(yīng)的濃度模式進行實驗測試,在該工藝中,tile在溶液中緩慢冷卻,使其能夠自組裝。作者使用熒光標(biāo)記來監(jiān)測每個形狀的組裝,并使用原子力顯微鏡對形狀進行成像。在 37 種提出的成核種子中,大約一半導(dǎo)致了所需形狀的選擇性自組裝。為了展示他們的系統(tǒng)的信息處理能力,作者使用它根據(jù)圖像中像素的陰影(灰度值)對 18 個 30×30像素的灰度圖像進行分類。這個想法是通過其中一個tile的濃度來表示每個圖像中每個像素的灰度值,以便生成的tile濃度模式促進指定形狀(H、A 或 M)的組裝。像素到tile的分配是通過計算完成的,以最大化指定形狀的自組裝,同時最小化競爭形狀的自組裝。至關(guān)重要的是,這項任務(wù)是針對所有圖像同時優(yōu)化的,而不是針對每個圖像獨立優(yōu)化的。當(dāng)作者通過實驗測試18個圖像的濃度模式時,他們觀察到所需的形狀確實比其他任何形狀更頻繁地組裝,其中13個圖像的選擇性超過80%。換句話說,tile系統(tǒng)通過組裝成指定的形狀來識別不同的濃度模式,從而識別相應(yīng)的圖像。至關(guān)重要的是,該系統(tǒng)還處理了12個圖像的降級版本。例如,當(dāng)馬圖像的某些像素的灰度值被隨機改變,從而破壞相應(yīng)的濃度模式時,系統(tǒng)仍然可靠地形成 H 形狀而不是 A 或 M,從而正確地對圖像進行分類。圖|模式識別的結(jié)果是一個預(yù)先存在的多種系統(tǒng)這項工作的主要限制之一是模式識別的速度、準(zhǔn)確性和復(fù)雜性之間的權(quán)衡。特別是,保守地選擇了實驗的時間尺度,以盡量減少不正確結(jié)構(gòu)的形成。贏者通吃的結(jié)果表明,這些時間尺度可以大大縮短;使用由較少tile組成的較小組件也可以加快速度。因為這樣的 DNA 系統(tǒng)可能會找到生物應(yīng)用,而不是成為基于硅的計算的替代品,所以速度考慮可能不如將計算直接嵌入納米級生物物理過程的能力重要。總的來說,最新的發(fā)現(xiàn)證明了如何在自組裝的生物物理過程中在分子水平上編碼復(fù)雜模式識別所需的計算。該研究還說明了先前關(guān)于 DNA-tile 組裝的理論和實驗工作如何支持復(fù)雜的新實驗的設(shè)計。此外,它還展示了DNA 的可編程性以及眾所周知的 DNA 堿基配對動力學(xué)和熱力學(xué)如何能夠設(shè)計具有 900 多個不同組件的自組裝系統(tǒng)來執(zhí)行復(fù)雜的計算。從計算的角度來看,未來工作的一個有希望的方向是進一步探索自組裝系統(tǒng)中的模式識別與其他形式的神經(jīng)計算之間的聯(lián)系。研究人員在之前的工作基礎(chǔ)上,發(fā)現(xiàn)了他們的tile系統(tǒng)和稱為 Hopfield 聯(lián)想記憶的神經(jīng)網(wǎng)絡(luò)模型,以及大腦中存儲空間記憶的位置細胞網(wǎng)絡(luò)之間的相似之處。進一步探索將神經(jīng)計算嵌入生物物理過程的機會和局限性將是有價值的。從實驗的角度來看,隨著科學(xué)家設(shè)計基于蛋白質(zhì)的系統(tǒng)和預(yù)測其生物物理相互作用的能力不斷提高,該研究概述的方法可用于設(shè)計處理信息的自組裝蛋白質(zhì)結(jié)構(gòu)。例如,具有不同功能的不同蛋白質(zhì)復(fù)合物可以根據(jù)其構(gòu)建模塊的濃度進行自組裝。更一般地說,這項工作還為未來設(shè)計嵌入生物物理過程的緊湊、穩(wěn)健和可擴展的計算提供了一個概念和實驗框架。Evans, C.G., O’Brien, J., Winfree, E. et al. Pattern recognition in the nucleation kinetics of non-equilibrium self-assembly. Nature 625, 500–507 (2024).https://doi.org/10.1038/s41586-023-06890-z