 通過蛋白質(zhì)-蛋白質(zhì)界面上精確設(shè)計的側(cè)鏈-側(cè)鏈相互作用來編程蛋白質(zhì)結(jié)晶極具挑戰(zhàn)性。在這里,華盛頓大學(xué)David Baker開發(fā)了一種通用的計算方法,用于以原子精度設(shè)計具有預(yù)先指定的晶格結(jié)構(gòu)的三維蛋白質(zhì)晶體。
通過蛋白質(zhì)-蛋白質(zhì)界面上精確設(shè)計的側(cè)鏈-側(cè)鏈相互作用來編程蛋白質(zhì)結(jié)晶極具挑戰(zhàn)性。在這里,華盛頓大學(xué)David Baker開發(fā)了一種通用的計算方法,用于以原子精度設(shè)計具有預(yù)先指定的晶格結(jié)構(gòu)的三維蛋白質(zhì)晶體。
本文要點:
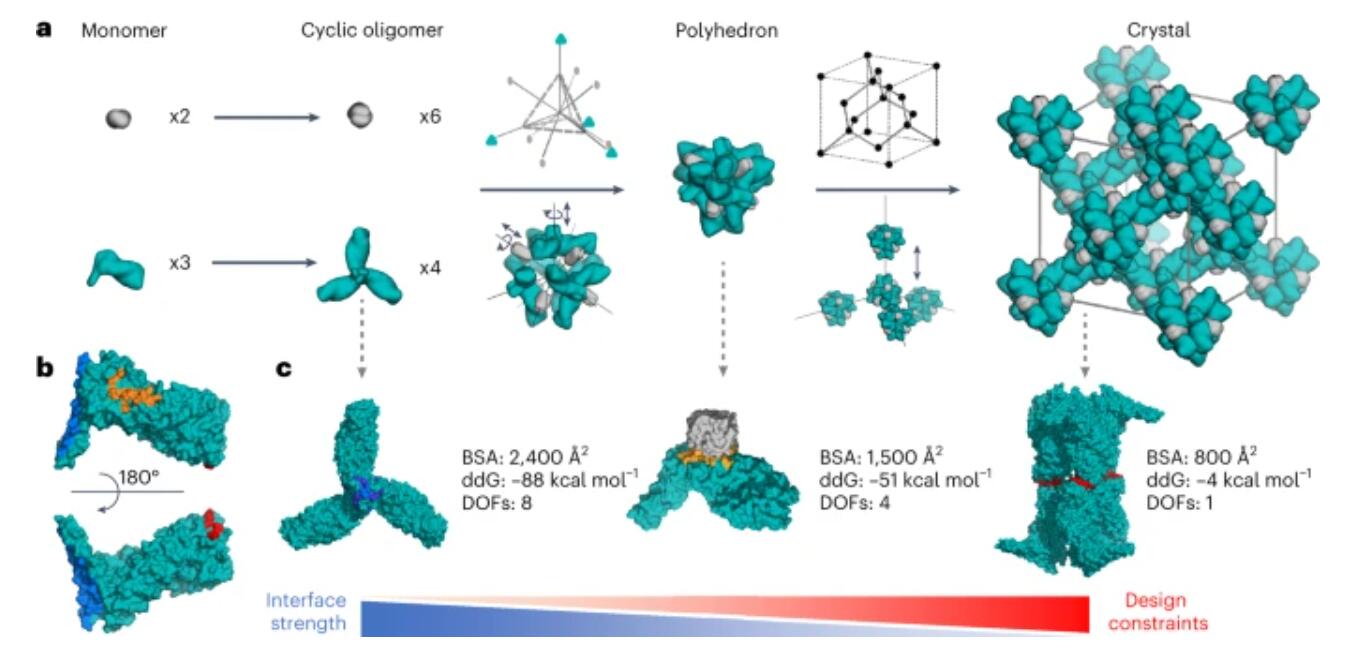
1) 作者設(shè)計了三對低聚物,它們可以單獨純化,混合后自發(fā)地自組裝成>100?μm三維晶體。這些晶體的結(jié)構(gòu)與計算設(shè)計模型幾乎相同,在整體結(jié)構(gòu)和特定的蛋白質(zhì)-蛋白質(zhì)相互作用方面都非常對應(yīng)。
2) 晶體晶胞的尺寸可以被系統(tǒng)地重新設(shè)計,同時保持空間群對稱性和整體結(jié)構(gòu),晶體具有極高的多孔性和高度穩(wěn)定性。該方法能夠高精度地進(jìn)行蛋白質(zhì)晶體的計算設(shè)計,所設(shè)計的蛋白質(zhì)晶體在其初級序列中編碼了結(jié)構(gòu)和組裝信息,為生物材料工程提供了強(qiáng)大的平臺。
Zhe Li et.al Accurate computational design of three-dimensional protein crystals Nature Materials 2023
DOI: 10.1038/s41563-023-01683-1
https://doi.org/10.1038/s41563-023-01683-1


















