
如何處理連續彎曲的分子是從低溫電子顯微鏡(cryo-EM)圖像中分析蛋白質單粒子的最大突出挑戰之一。鑒于此,來自劍橋大學MRC分子生物實驗室的Sjors H. W. Scheres、Johannes Schwab等人開發了一種軟件工具DynaMight。
文章要點:
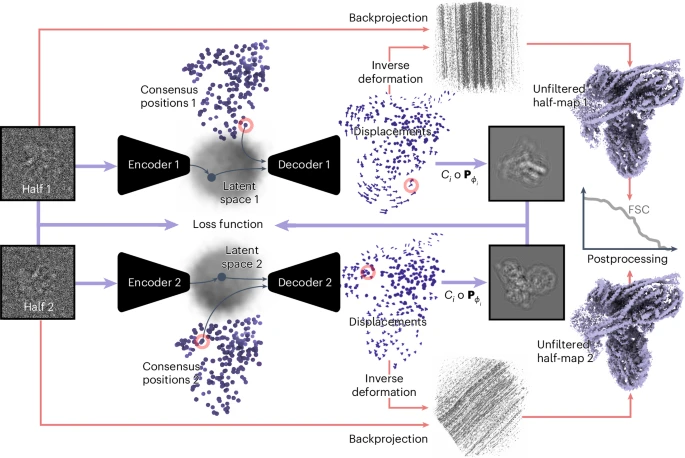
1) 該研究開發的這種工具是通過學習每個粒子圖像的共識結構的高斯偽原子模型的三維變形,以此來估計低溫EM數據集中的連續構象空間;
2) 此外,該研究還還展示了如何通過在半組冷凍EM數據上獨立訓練兩個變分自編碼器來獲得變形的誤差估計,以及通過使用原子模型對三維變形進行正則化可能會因模型偏差而導致重要偽影,DynaMight作為RELION-5的一部分作為免費開源軟件分發。
參考資料:
Schwab, J., Kimanius, D., Burt, A. et al. DynaMight: estimating molecular motions with improved reconstruction from cryo-EM images. Nat Methods (2024).
10.1038/s41592-024-02377-5
https://doi.org/10.1038/s41592-024-02377-5


















